放弃Venn-Upset-花瓣图,拥抱二分网络
写在前面
让点随机排布在一个区域,保证点之间不重叠,并且将点的图层放到最上层,保证节点最清晰,然后边可以进行透明化,更加突出节点的位置。这里我新构建了布局函数 PolyRdmNotdCirG 来做这个随机排布。调用的是packcircles包的算法。使用和其他相似函数一样,这里我们重点介绍一下使用这种算法构造的二分网络布局。
微生物网络
ggClusterNet 安装
ggClusterNet包依赖的R包均在cran或者biocductor中,所以未能成功安装,需要检查依赖是否都顺利安装。如果网路问题,无法下载R包,可以在github中手动下载安装
#---ggClusterNet
devtools::install_github("taowenmicro/ggClusterNet")
#--如果无法安装请检查网络或者换个时间
导入R包和输入文件
#--导入所需R包#-------
library(ggplot2)
library(ggrepel)
library(ggClusterNet)
library(phyloseq)
library(dplyr)# 数据内置
#-----导入数据#-------
data(ps)#--可选
#-----导入数据#-------
ps = readRDS("../ori_data/ps_liu.rds")
这里我们提取一部分OTU,节省出图时间。
# ps
data(ps)ps_sub = filter_taxa(ps, function(x) sum(x ) > 20 , TRUE)
ps_sub = filter_taxa(ps_sub, function(x) sum(x ) < 30 , TRUE)
ps_sub
div_network函数 用于计算共有和特有关系
这个函数是之前我写的专门用于从OTU表格整理成Gephi的输入文件,所以大家直接用这个函数即可转到gephi进行操作。这次为了配合二分网络,我设置了参数flour = TRUE,代表是否仅仅提取共有部分和特有部分。
# ?div_network
result = div_network(ps_sub,num = 6)edge = result[[1]]
head(edge)# levels(edge$target)
# node = result[[2]]
# head(node)
#
# tail(node)
data = result[[3]]
dim(data)#----计算节点坐标
# flour参数,设置是否仅仅展示共有和特有的二分网络
div_culculate函数 核心算法,用于计算二分网络的节点和边的表格
参数解释:
distance = 1.1:
中心一团点到样本点距离
distance2 = 1.5:
中心点模块到独有OTU点之间距离
distance3 = 1.3:
样本点和独有OTU之间的距离
order = FALSE :
节点是否需要随机扰动效果
result <- div_culculate(table = result[[3]],distance = 1.1,distance2 = 1.5,distance3 = 1.3,order = FALSE)edge = result[[1]]
head(edge)plotdata = result[[2]]
head(plotdata)
#--这部分数据是样本点数据
groupdata <- result[[3]]
对OTU进行注释,方便添加到图形上
为了让节点更加丰富,这里我对节点文件添加了注释信息。
# table(plotdata$elements)
node = plotdata[plotdata$elements == unique(plotdata$elements), ]otu_table = as.data.frame(t(vegan_otu(ps_sub)))
tax_table = as.data.frame(vegan_tax(ps_sub))
res = merge(node,tax_table,by = "row.names",all = F)
dim(res)
head(res)
row.names(res) = res$Row.names
res$Row.names = NULL
plotcord = resxx = data.frame(mean =rowMeans(otu_table))
head(xx)
plotcord = merge(plotcord,xx,by = "row.names",all = FALSE)
head(plotcord)
# plotcord$Phylum
row.names(plotcord) = plotcord$Row.names
plotcord$Row.names = NULL
head(plotcord)
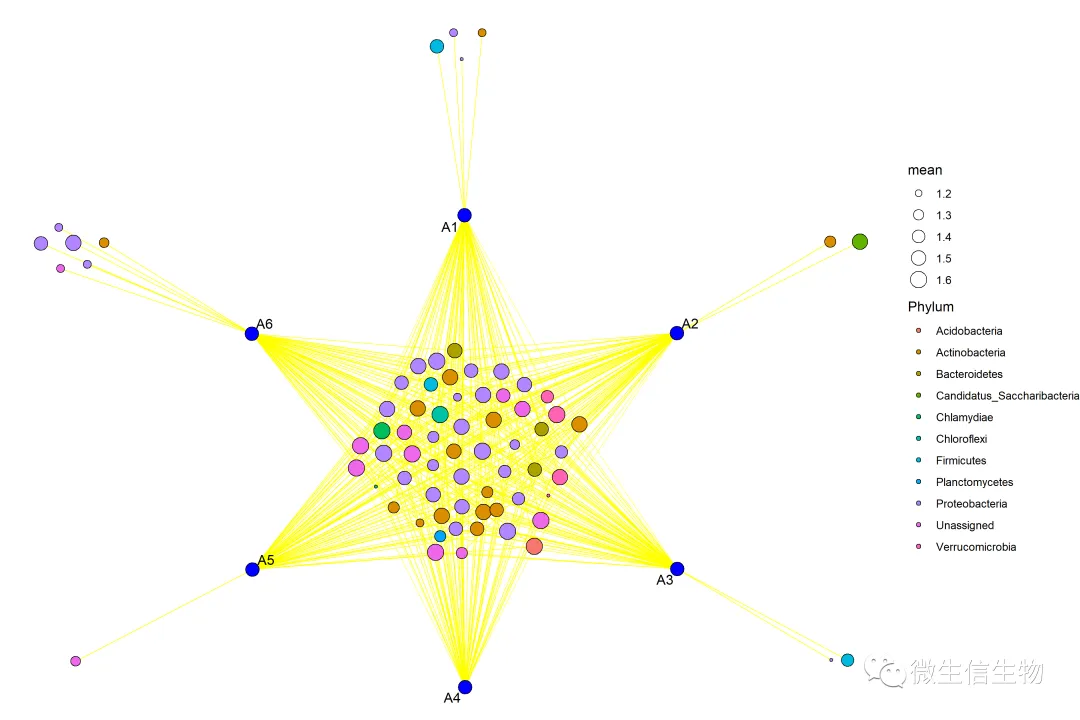
p = ggplot() + geom_segment(aes(x = X1, y = Y1, xend = X2, yend = Y2),data = edge, size = 0.3,color = "yellow") +geom_point(aes(X1, X2,fill = Phylum,size =mean ),pch = 21, data = plotcord) +geom_point(aes(X1, X2),pch = 21, data = groupdata,size = 5,fill = "blue",color = "black") +geom_text_repel(aes(X1, X2,label = elements ), data = groupdata) +theme_void()pggsave("4.png",p,width = 12,height = 8)

map = as.data.frame(sample_data(ps_sub))map$Group2 <- rep(c("A1","A2","A3","A4","A5","A6"),3)sample_data(ps_sub) <- map
# ?div_network
result = div_network(ps_sub,num = 3,group = "Group2",flour = TRUE)edge = result[[1]]
head(edge)# levels(edge$target)
# node = result[[2]]
# head(node)
#
# tail(node)data = result[[3]]
dim(data)#----计算节点坐标
# flour参数,设置是否仅仅展示共有和特有的二分网络result <- div_culculate(table = result[[3]],distance = 1.1,distance2 = 1.5,distance3 = 1.3,order = FALSE)edge = result[[1]]
head(edge)plotdata = result[[2]]
head(plotdata)groupdata <- result[[3]]# table(plotdata$elements)
node = plotdata[plotdata$elements == unique(plotdata$elements), ]otu_table = as.data.frame(t(vegan_otu(ps_sub)))
tax_table = as.data.frame(vegan_tax(ps_sub))
res = merge(node,tax_table,by = "row.names",all = F)
dim(res)
head(res)
row.names(res) = res$Row.names
res$Row.names = NULL
plotcord = resxx = data.frame(mean =rowMeans(otu_table))
head(xx)
plotcord = merge(plotcord,xx,by = "row.names",all = FALSE)
head(plotcord)
# plotcord$Phylum
row.names(plotcord) = plotcord$Row.names
plotcord$Row.names = NULL
head(plotcord)p = ggplot() + geom_segment(aes(x = X1, y = Y1, xend = X2, yend = Y2),data = edge, size = 0.3,color = "yellow") +geom_point(aes(X1, X2,fill = Phylum,size =mean ),pch = 21, data = plotcord) +geom_point(aes(X1, X2),pch = 21, data = groupdata,size = 5,fill = "blue",color = "black") +geom_text_repel(aes(X1, X2,label = elements ), data = groupdata) +theme_void()
p
ggsave("4.png",p,width = 12,height = 8)
map = as.data.frame(sample_data(ps_sub))map = map[1:12,]# map$Group2 <- rep(c("A1","A2","A3","A4","A5","A6"),2)
sample_data(ps_sub) <- mapresult = div_network(ps_sub,num = 3,group = "Group",flour = TRUE)edge = result[[1]]
head(edge)# levels(edge$target)
# node = result[[2]]
# head(node)
#
# tail(node)data = result[[3]]
dim(data)result <- div_culculate(table = result[[3]],distance = 1.1,distance2 = 1.5,distance3 = 1.3,order = FALSE)edge = result[[1]]
head(edge)plotdata = result[[2]]
head(plotdata)groupdata <- result[[3]]# table(plotdata$elements)
node = plotdata[plotdata$elements == unique(plotdata$elements), ]otu_table = as.data.frame(t(vegan_otu(ps_sub)))
tax_table = as.data.frame(vegan_tax(ps_sub))
res = merge(node,tax_table,by = "row.names",all = F)
dim(res)
head(res)
row.names(res) = res$Row.names
res$Row.names = NULL
plotcord = resxx = data.frame(mean =rowMeans(otu_table))
head(xx)
plotcord = merge(plotcord,xx,by = "row.names",all = FALSE)
head(plotcord)
# plotcord$Phylum
row.names(plotcord) = plotcord$Row.names
plotcord$Row.names = NULL
head(plotcord)p = ggplot() + geom_segment(aes(x = X1, y = Y1, xend = X2, yend = Y2),data = edge, size = 0.3,color = "yellow") +geom_point(aes(X1, X2,fill = Phylum,size =mean ),pch = 21, data = plotcord) +geom_point(aes(X1, X2),pch = 21, data = groupdata,size = 5,fill = "blue",color = "black") +geom_text_repel(aes(X1, X2,label = elements ), data = groupdata) +theme_void()p# ggsave("4.png",p,width = 12,height = 22)